Ribosome
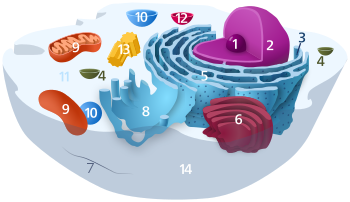
Schéma d'une cellule animale type. Organites :
(1) Nucléole
(2) Noyau
(3) Ribosomes
(4) Vésicule
(5) Réticulum endoplasmique rugueux (ou granuleux) (REG)
(6) Appareil de Golgi
(7) Cytosquelette
(8) Réticulum endoplasmique lisse
(9) Mitochondries
(10) Vacuole (absent des cellules animales)
(11) Cytosol
(12) Lysosome
(13) Centrosome (constitué de deux centrioles)
(14) Membrane plasmique
Les ribosomes sont des complexes ribonucléoprotéiques (c'est-à-dire composés de protéines et d'ARN) extrêmement conservés au cours de l'évolution présents dans les cellules eucaryotes et procaryotes. Leur fonction est de synthétiser les protéines en décodant l'information contenue dans l'ARN messager. Ils sont constitués d'ARN ribosomiques, qui portent l'activité catalytique, et de protéines ribosomiques. Les ribosomes sont constitués de deux sous-unités, une plus petite qui « lit » l'ARN messager et une plus grosse qui se charge de la polymérisation des acides aminés pour former la protéine correspondante.
Sommaire
1 Localisation
2 Structure
3 Fonctions
4 Découvertes
5 Notes et références
6 Voir aussi
6.1 Bibliographie
6.2 Articles connexes
6.3 Liens externes
Localisation |
Chez les eucaryotes, les ribosomes se trouvent dans le cytoplasme, libres, ou associés, soit aux membranes du réticulum endoplasmique, soit à l'enveloppe nucléaire. Chez les bactéries, on trouve aussi les ribosomes dans le cytoplasme ou liés à la membrane plasmique. On trouve aussi des ribosomes dans les mitochondries et certains plastes, et leur structure est proche de celle des ribosomes procaryotes. Ceci s'explique par la théorie de l'endosymbiose. Leur fonction est de traduire les ARN messagers issus de l'ADN mitochondrial ou de l'ADN chloroplastique.
Lorsque la traduction est très active, plusieurs ribosomes peuvent être associés simultanément à un même ARN messager, ce chapelet de ribosomes consécutifs sur l'ARN étant appelé polysome ou polyribosome.
Structure |

Structure atomique de la grande sous-unité 50S du ribosome d'une cellule procaryote.
Les protéines sont colorées en bleu et les ARN en brun. Le site actif, l'adénine 2486, est coloré en rouge.
Comportant des ARN dits ARN ribosomiques (ou ARNr) et des protéines ribosomiques, ils sont composés de deux sous-unités : une grande (L pour large) et une petite (S pour small) sous-unité. Ces sous-unités sont construites autour d'un cœur d'ARN ribosomique possédant une structure très compacte, autour duquel sont accrochées les protéines. Le site actif du ribosome qui catalyse la formation de liaison peptidique est constitué d'ARN. Chez les eucaryotes, la biogenèse des ribosomes a lieu dans le nucléole, une structure interne du noyau.
- Grande sous-unité : dans le ribosome cytoplasmique des eucaryotes, elle est constituée de trois molécules d'ARNr (5S, 28S et 5,8S, comportant respectivement environ 120, 4 700 et 160 nucléotides), et de 49 protéines ribosomiques. Cette grande sous-unité a une masse moléculaire de 2,8.106daltons, un coefficient de sédimentation de 60 svedbergs. Chez les procaryotes, cette grande sous-unité est caractérisée par un coefficient de sédimentation de 50 svedbergs ; elle est composée d'ARN 23S (2 900 nucléotides[1]), d'ARN 5S (120 nucléotides) et de 34 protéines.
- Petite sous-unité : dans le ribosome cytoplasmique des eucaryotes, elle n'est constituée que d'une molécule d'ARNr (18S, comportant 1 900 nucléotides) et de 33 protéines ribosomiques. Cette petite sous-unité a une masse moléculaire de 1,4.106 daltons, un coefficient de sédimentation de 40 svedbergs. Chez les procaryotes, elle est constituée d'ARNr 16S (1 540 nucléotides[2]) et de 21 protéines. Son coefficient de sédimentation est de 30 svedbergs.
Au total, le ribosome fonctionnel (composé des deux sous-unités réunies) a une masse moléculaire de 4,2.106 daltons, un coefficient de sédimentation de 80 svedbergs chez les eucaryotes et 70 svedbergs chez les procaryotes.
Les ribosomes 70S des procaryotes ont une taille de 25 nanomètres de diamètre (il y a une distance de 20 nanomètres entre le site A et le site E).
Les ribosomes 70S des procaryotes sont sensibles à certains antibiotiques (amino-glycosides et chloramphénicol par exemple) qui n'ont pas d'effet sur les ribosomes 80S des eucaryotes.
Les mitochondries et les chloroplastes (des cellules végétales) contiennent également des ribosomes 70S, ce qui, en plus du fait qu'ils ont leur propre ADN et leur propre mécanisme de reproduction, étayerait la thèse selon laquelle ces organites seraient issus de procaryotes en symbiose avec la cellule eucaryote (théorie de l'endosymbiose).
Fonctions |
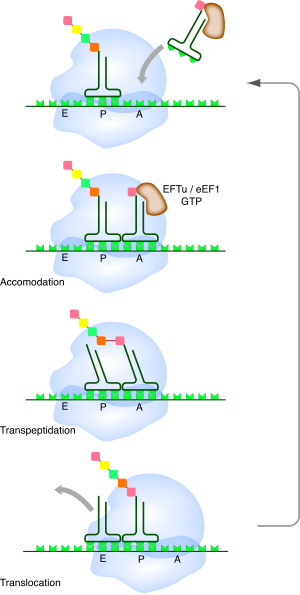
Processus de traduction de l'ARN en protéine par le ribosome.
Le ribosome est la « machine » qui assure la traduction de la molécule d'ARNm dans la synthèse des protéines. Le code génétique assure la correspondance entre la séquence de l'ARNm et la séquence du polypeptide synthétisé. Le ribosome utilise les ARN de transfert ou ARNt comme « adaptateurs » entre l'ARN messager et les acides aminés.
L'ARN messager passe à travers la petite sous-unité (30S ou 40S) qui contient les sites de fixation des ARNt sur l'ARNm. La grande sous-unité contient la partie catalytique, appelée centre peptidyl-transférase, qui effectue la synthèse de la liaison peptidique entre les acides aminés consécutifs de la protéine. La grande sous-unité contient également un tunnel par lequel sort la chaîne protéique en cours de synthèse. Il existe aussi dans la grande sous-unité trois sites où vont se fixer les ARNt porteurs des acides aminés pendant la traduction : le site A (pour aminoacyl-ARNt), qui est occupé par un ARNt porteur d'un acide aminé en attente d'être lié à la chaîne polypeptidique ; le site P (pour peptidyl-ARNt), qui est occupé par un ARNt porteur d'un acide aminé lié à la chaîne polypeptidique résultante ; enfin, le site E (pour exit), qui permet la libération de l'ARNt désacétylé qui a livré son acide aminé.
Le ribosome est de plus un moteur moléculaire, qui avance sur l'ARN messager en consommant l'énergie fournie par l'hydrolyse de la GTP. Plusieurs protéines, appelées facteurs d'élongation, sont associées à ce mouvement, appelé translocation.
Découvertes |
En février 2009, la revue Nature a publié un article rédigé par des biophysiciens de l'Université de Montréal : ils y expliquent les mécanismes de formation de cette molécule qui peut contenir jusqu'à 300 000 atomes[3]. Ces explications permettraient de mieux comprendre les mécanismes d'émergence des êtres vivants.
Notes et références |
J. Brosius, T. J. Dull et H. F. Noller, « Complete nucleotide sequence of a 23S ribosomal RNA gene from Escherichia coli », Proceedings of the National Academy of Sciences of the United States of America, vol. 77, 1er janvier 1980, p. 201–204 (ISSN 0027-8424, lire en ligne, consulté le 15 octobre 2016)
J. Brosius, M. L. Palmer, P. J. Kennedy et H. F. Noller, « Complete nucleotide sequence of a 16S ribosomal RNA gene from Escherichia coli », Proceedings of the National Academy of Sciences of the United States of America, vol. 75, 1er octobre 1978, p. 4801–4805 (ISSN 0027-8424, lire en ligne, consulté le 15 octobre 2016)
Pauline Gravel, « Découverte du chaînon manquant aux origines de la vie », Le Devoir, 21 février 2009(lire en ligne)
Voir aussi |
Bibliographie |
- D. Fourmy, S. Yoshizawa et H. Grosjean, « Le ribosome : l'usine à protéines », Pour la Science, 313, novembre 2003.
Articles connexes |
- Biosynthèse des protéines
- ARNm
- ARNr
- ARNt
- Code génétique
- Protéine
- Ribozyme
Liens externes |
(en) Vidéo de reconstitutions en 3 dimensions de la structure et du mode d'action des ribosomes, sur le site de Tom Steitz, récipiendaire du prix Nobel de chimie 2009
- Portail de la biologie cellulaire et moléculaire
